


背景说明
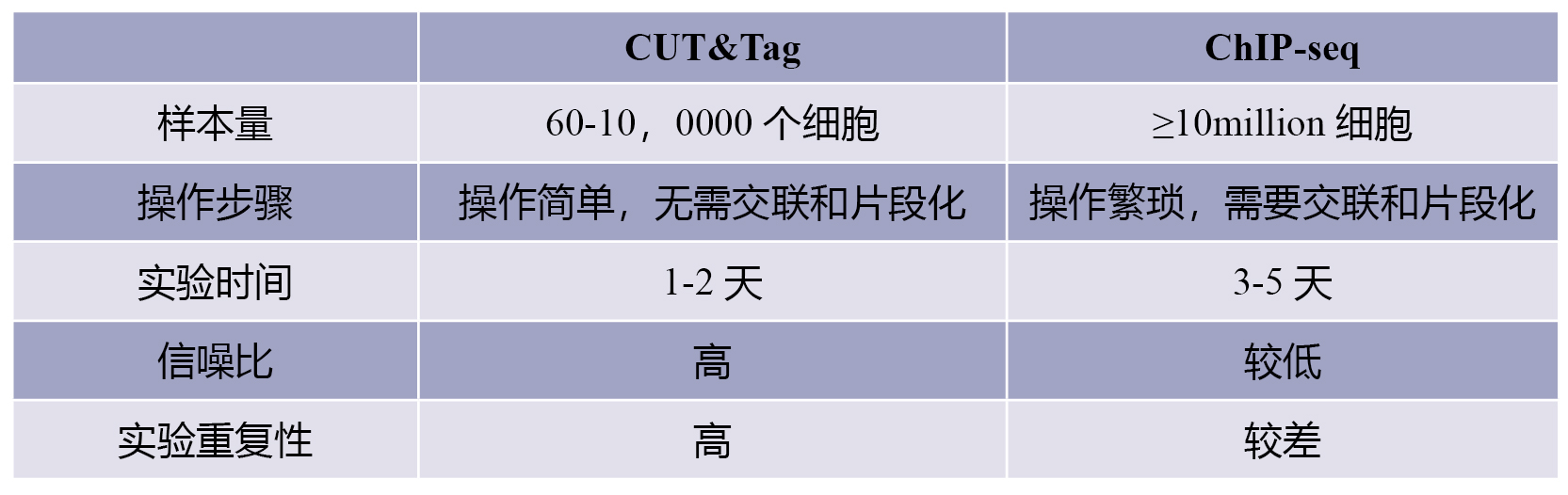
在ChIP中,超声波随机打断的染色质,经过抗体免疫沉淀下来的DNA,通常长度为几百至几千bp。然而,在CUT&Tag中,转座子只在接近蛋白质结合位点的附近切割染色质,从而使DNA序列长度缩短。因此即使较低的测序深度(3-5M)也能得到高质量的数据,并且其背景信号比大多数ChIP-seq的背景信号低。
原理简介
CUT&Tag技术的核心是在Tn5上融合了protein A/G抗体结合功能域的转座体pAG-Tn5。在进行CUT&Tag实验时,首先进行靶蛋白特异性抗体(一抗)孵育,使抗体进入细胞与靶蛋白结合。为了放大信号,同理接着进行二抗孵育。最后孵育pAG-Tn5转座体,使得转座体进入细胞并与抗体结合,这样就把转座体间接的固定在靶蛋白上,随后加入Mg2+,激活Tn5酶的切割活性,切断靶蛋白结合的DNA区域。由于Tn5连有测序接头,在打断的同时直接在片段化的DNA上加接头,接着提取DNA,进行PCR扩增构建文库。
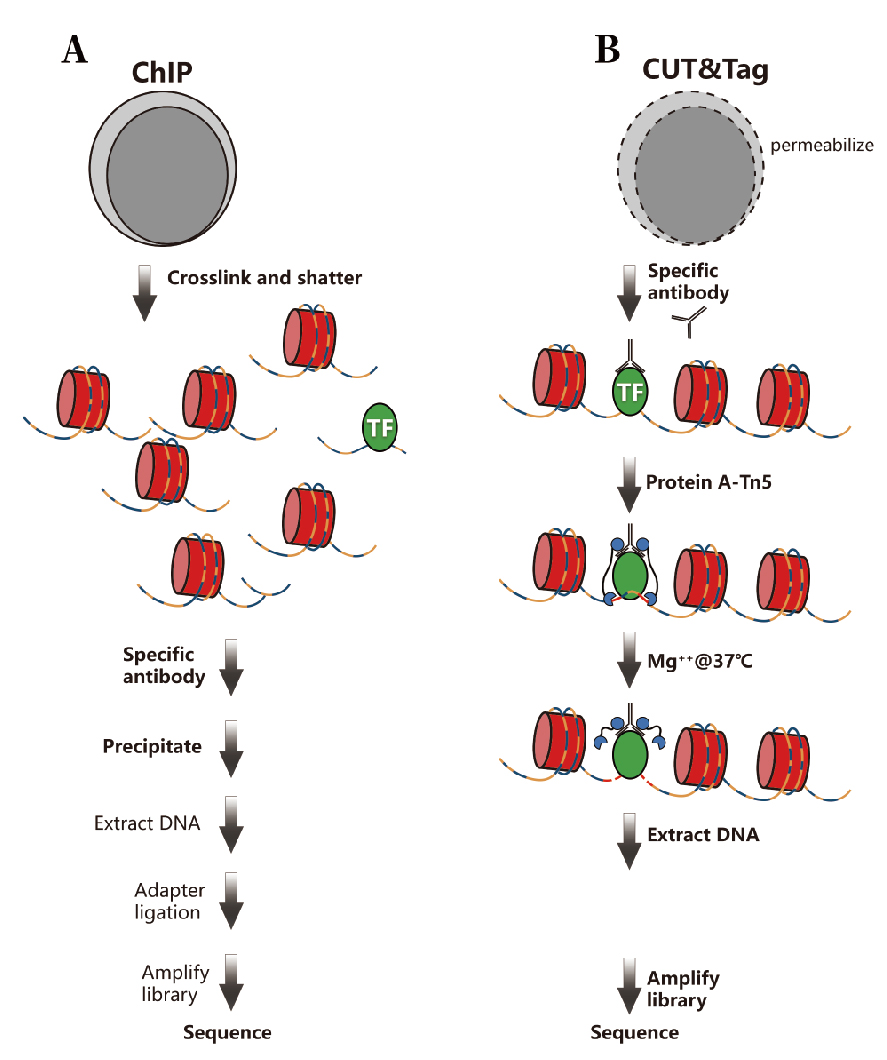
图 ChIP-Seq与CUT&Tag实验程序比较(Zheng et al., 2020)。
尽管CUT&Tag与ChIP方法在某些方面类似,但CUT&Tag实验的起始材料是活的可渗透细胞或分离的细胞核,而不是ChIP-seq中使用的甲醛交联的细胞或组织。CUT&Tag有望将蛋白与染色质DNA互作的研究变成了一种类似PCR反应的常规操作,对基因调控、表观遗传等领域的研究具有重要的意义。
表 CUT&Tag与ChIP-seq比较
服务流程
客户在线下单——订单/实验材料确认——细胞培养——抗体孵育——pA/pG-Tn5转座子孵育——转座子激活——DNA片段化——DNA提取——文库扩增与纯化——高通量测序——数据分析——结果交付
服务内容及说明
适用范围
1、用于表观遗传学领域的蛋白质与DNA互作研究;
2、研究转录因子在全基因组上的结合或分布位点;
3、研究组蛋白修饰在全基因组上的分布位点;
4、寻找转录因子下游调控的靶基因。
实验周期
致电详询
客户下单及项目信息填写
在我司官网http://www.biorun.com/进行注册或登录,请客户按照页面提示填写项目名称、选择项目类型、填写个人信息及联系方式,提交项目所需要的具体信息,包括:
1、组织、细胞、DNA等样品来源、状态及其他相关信息;
2、尽可能丰富的相关资料、文献。
实验信息
实验过程中客户可以随时登入管理系统查看项目实时进展情况。实验结题时系统会通过短信自动通知客户,并发送实验报告查看网址。实验结题后,实验报告,检测结果可在线查看或打印,并永久保留。
实验交付内容
1、所有实验的原始数据(包括实验过程、实验试剂与设备等);
2、表观组学基础数据分析与差异数据分析(生物信息学分析);
3、生物信息学分析内容包括:原始数据质控、参考基因组比对、Peak基因注释、峰图分析及注释、Motif分析、GO注释分析、KEGG注释分析等。
文献示例
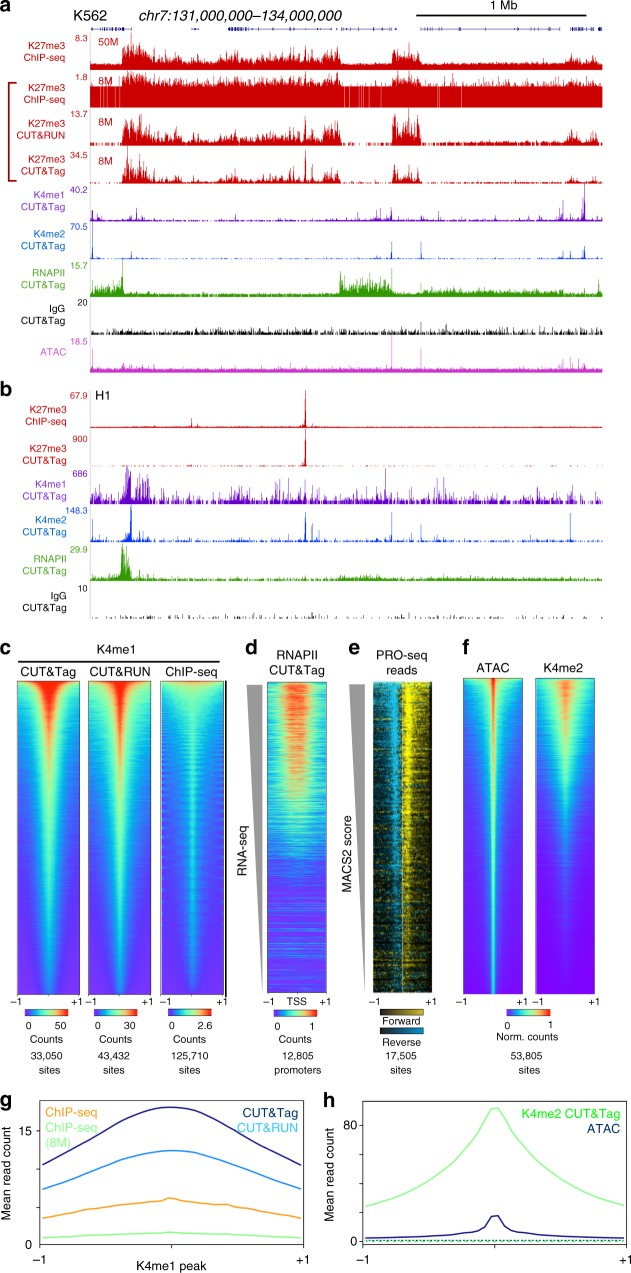
图 CUT&Tag用于组蛋白修饰分析和RNAPII(Kaya-Okur et al., 2019)。
参考文献
Ye Zheng, Kami Ahmad, Steven Henikoff. CUT&Tag Data Processing and Analysis Tutorial. Protocol .io, 2020.
Kaya-Okur HS, Wu SJ, Codomo CA, et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019;10(1):1930.