

背景说明
真核生物的基因通常是由多个外显子(编码区)和内含子(非编码区)交替组成。剪接是一个复杂的过程,它会移除前体mRNA(pre-mRNA)中的内含子,连接外显子,最终形成成熟的mRNA,用于蛋白质翻译。错误的剪接会导致mRNA和蛋白质异常,与多种遗传疾病相关。剪接过程是由一种高分子量的剪接体介导完成的。剪接体是由U1、U2、U4、U5和U6五种小核糖核酸蛋白(Small nuclear ribonucleoprotein, snRNPs)和多种非snRNP蛋白质组成的一种复合体。snRNPs和其他复合体蛋白质相互作用,形成功能复杂的结构,确保剪接体能够正确地定位和识别剪接位点,完成剪接过程。
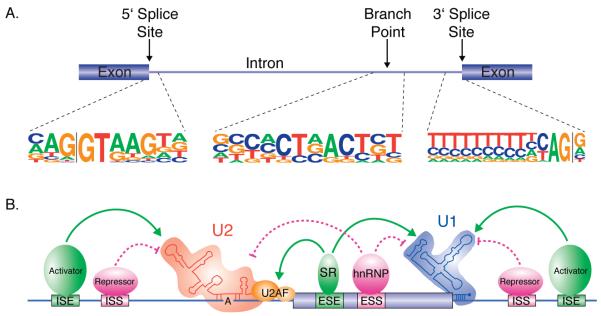
图 选择性剪接的经典过程(McManus et al., 2011)。
minigene实验以微型报告基因为核心,模拟体内可变剪接,将变异位点附近的外显子和内含子插入到minigene载体MCS区域构建报告质粒,该报告质粒除常规启动子和终止子元件外,还包含剪接供体、剪接受体等元件,形成微型转录单位。质粒转染培养细胞后,RNA聚合酶Ⅱ启动转录,剪接复合物按细胞环境识别待测序列,剪接后产生mRNA。转染24 h后提取RNA,通过RT-PCR、DNA电泳及测序分析转录产物。最后比较野生型与突变型的RNA剪接差异,解析突变(如SNV、Indel)如何改变RNA的剪接导致遗传病,是验证剪接调控元件功能、药物干预mRNA剪接的经典技术。
服务内容及说明
适用范围
(1)疾病机制研究
①遗传病/罕见病的发病机制
minigene系统可快速构建患者的特异性突变(如5'或3'剪接位点突变、分支位点突变、外显子沉默子/增强子突变)的微型基因模型,在细胞水平重现疾病相关剪接异常。
②癌症的发生机制
将癌症相关基因的(如BRCA1、TP53)异常剪接区域克隆至minigene报告载体,在肿瘤细胞系中监测突变所导致的mRNA剪接变化。
(2)药物开发
反义寡核苷酸(ASO)通过碱基互补配对靶向pre-mRNA特定区域,可精确调控mRNA剪接;而minigene系统则能快速构建包含目标剪接位点的报告基因,用于高效验证ASO效果或筛选小分子剪接调节剂。
(3)基础科研
①剪接调控因子功能鉴定,通过突变关键剪接位点或调控元件(如ESE/ESS),鉴定新型剪接因子(如SRSF、hnRNP家族蛋白)的作用机制。
②进化保守性分析,比较不同物种(如人、小鼠、斑马鱼)的同源基因minigene剪接模式,揭示剪接调控的进化选择压力。
技术优势
1、高效灵活
无需全长基因,仅构建包含目标外显子+侧翼内含子关键区域(通常200 - 300 bp)的微型基因载体,而非全长基因(可能长达数kb),就能够在体外模拟pre-mRNA剪接环境,实现在体外对剪接事件进行验证。
2、精准可控
精准构建剪接位点或调控序列(如剪接因子结合位点)野生型及突变型载体,载体背景统一,转染剂量、细胞环境可控,实现变量单一化,结果直接归因于目标序列,实验结果重复性高。
3、广谱适用
不依赖物种基因组大小,可在人、鼠等多物种细胞中瞬时表达;兼容多种突变类型及药物处理,适配遗传病、癌症、药物筛选等广谱研究场景。
客户下单及项目信息填写
在我司官网http://www.biorun.com/进行注册或登录,请客户按照页面提示填写:样本来源、检测指标、样本类型、检测方式等信息,检测试剂盒可选自行提供或我司提供。
实验信息
实验过程中客户可以随时登入管理系统查看项目实时进展情况。实验结题时系统会通过短信自动通知客户,并发送实验报告查看网址。实验结题后,实验报告、检测结果可在线查看或打印,并永久保留。
实验周期
致电详询
实验交付内容
①载体构建图谱及测序结果;
②一份去内毒素质粒,浓度约500 ng/μL,质量10 μg;一份大肠杆菌菌液1 mL(25%甘油菌);
③剪接结果分析,原始数据及结果报告。
文献案例
1、在罕见病研究中的应用
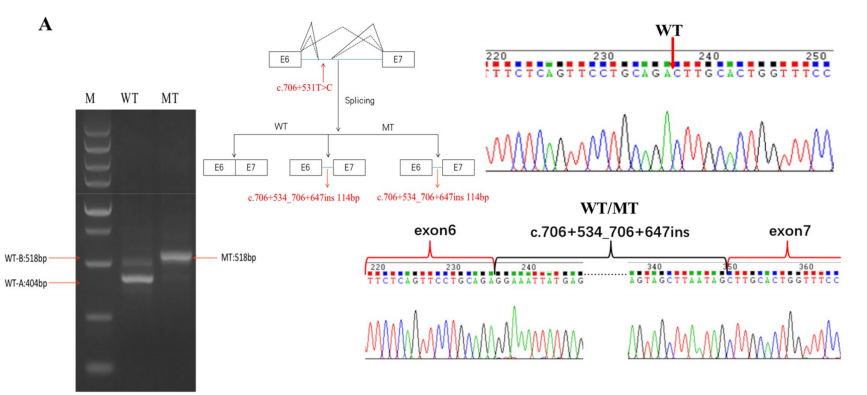
苯丙酮尿症患者中携带PAH基因的点突变c.706+531T>C,导致PAH基因6号外显子下游插入114 bp序列(Zhang et al., 2023)。
2、在肿瘤研究中的应用
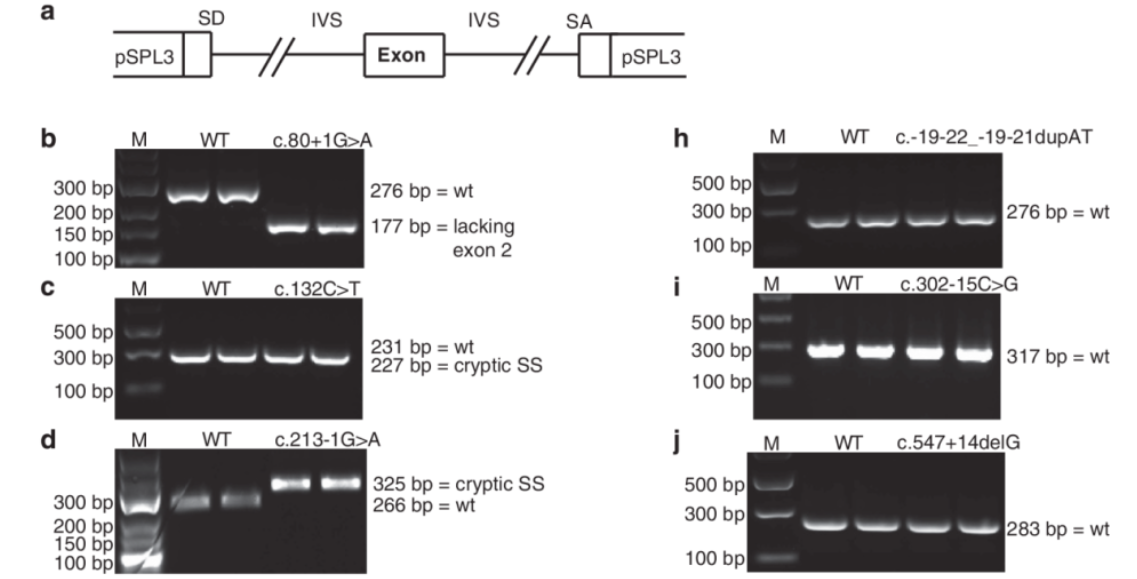
BRCA1基因位点突变会增加乳腺癌和卵巢癌的得病风险,BRCA1基因的突变位点对剪接改变的验证(Steffensen at al., 2014)。
3、在基础科研中应用
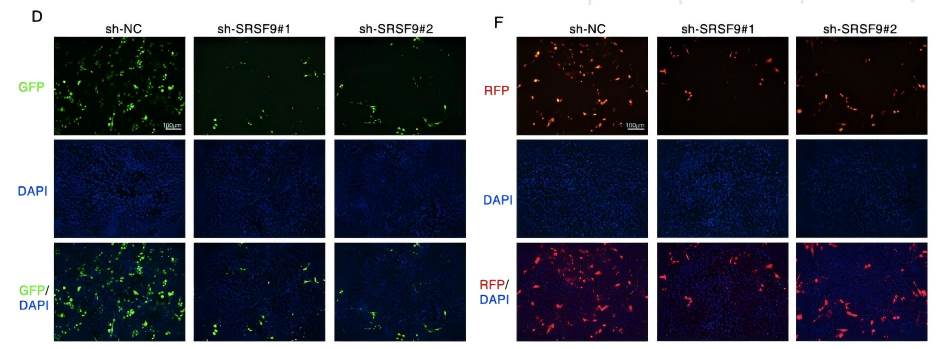
研究者Peng等人构建了一个EGFP/RFP-minigene系统,可以通过荧光来检测外显子的跳跃,验证了剪接因子SRSF9的存在会抑制SLC37A4第7号外显子的跳跃(Peng et al., 2025)。
4、在药物筛选中的应用
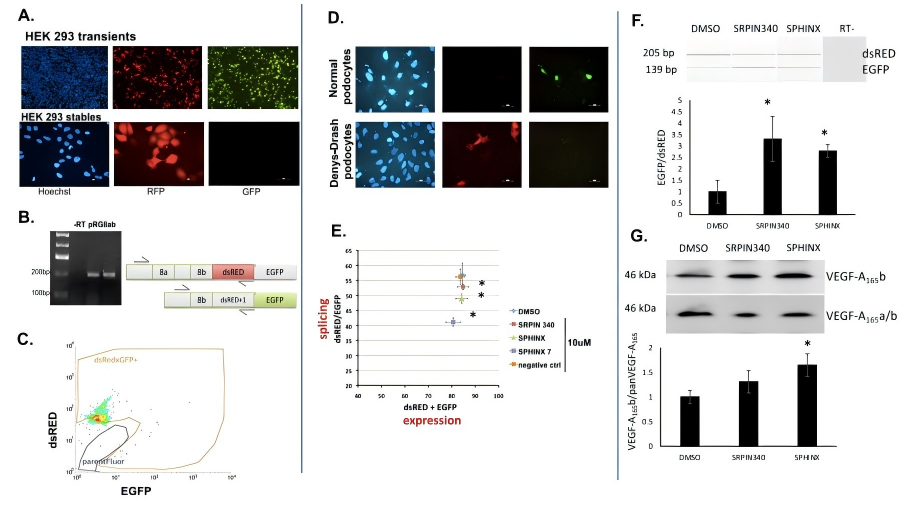
双荧光-minigene系统把“RNA剪接事件”翻译成“红/绿光信号”,已成为解析可变剪接调控网络及发现剪接靶向药物的有力工具(Star E and Stevens M., 2021)。
参考文献
Peng Q, Wang L, Long Y, et al. SRSF9 mediates oncogenic RNA splicing of SLC37A4 via liquid–liquid phase separation to promote oral cancer progression[J]. Journal of Advanced Research, 2025.
Star E, Stevens M,Gooding C, et al. A drug-repositioning screen using splicing-sensitive fluorescent reporters identifies novel modulators of VEGF-A splicing with anti-angiogenic properties[J]. Oncogenesis,2021,10(5): 36.
Steffensen A Y, Dandanell M, Jønson L, et al. Functional characterization of BRCA1 gene variants by mini-gene splicing assay[J]. European Journal of Human Genetics, 2014, 22(12): 1362-1368.
Zhang C, Yan Y, Zhou B, et al. Identification of deep intronic variants of PAH in phenylketonuria using full-length gene sequencing[J]. Orphanet journal of rare diseases, 2023, 18(1): 128.
Zhu Y, Wu K, Wen H. Functional analysis of a novel homozygous missense IVD gene variant: a case report with dual genetic diagnoses[J]. Frontiers in Pediatrics, 2025, 13: 1494530.