

背景说明
一个作物有多个栽培种或生态型,也存在一些本地种和野生种。不同品系之间存在大量的DNA变异,包括SNP、InDel、PAV、CNV等。基因组重测序是利用超声或者酶切等方法,将DNA片段化,再通过高通量测序的方法检测全基因组序列,然后利用生物信息学的方法将DNA变异解析出来的方法。
服务内容及说明
1、基础分析(包括SNP和InDel等)。
2、个性化分析服务内容:
3、系统进化树分析,分析样本间的进化关系;
4、群体主成分分析,分析群体的遗传变异分布结构;
5、连锁不平衡分析,分析不同位点间的连锁遗传程度;
6、单倍型图谱分析,分析共遗传的等位基因型组合;
7、全基因组关联分析,分析与性状关联的SNP或Indel;
8、性状关联区域基因注释,分析目标区域的基因和基因功能;
9、eQTL分析,分析与目标基因RNA表达调控关联的遗传变异;
10、pQTL分析,分析与目标基因蛋白表达调控关联的遗传变异;
11、mQTL分析,分析与代谢物变化关联的遗传变异;
12、HaWAS分析,分析与目标区域可及性变化关联的遗传变异;
服务周期
1、两周交付基础分析结果。
2、个性化分析的周期需要根据客户需要做的分析项目来定,具体的服务周期可直接与区域销售经理联系。
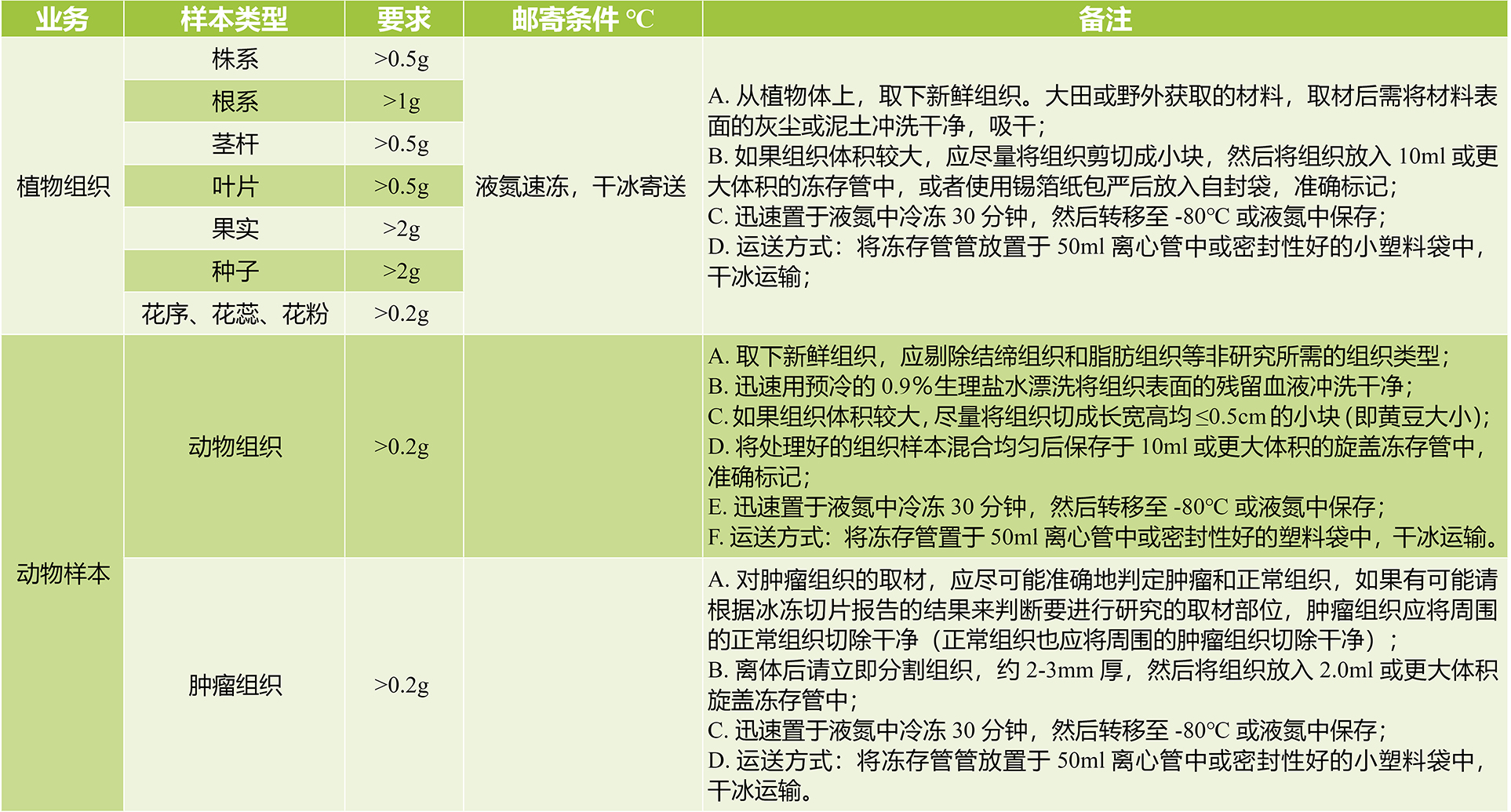
收样标准
客户下单及项目信息填写
在我司官网http://www.biorun.com/进行注册或登录,请客户按照页面提示填写项目名称、选择项目类型、填写个人信息及联系方式,提交项目所需要的具体信息,包括:
1、样品来源、状态、物种及其他相关信息;
2、尽可能丰富的相关资料、文献。
实验信息
实验过程中客户可以随时登入管理系统查看项目实时进展情况。实验结题时系统会通过短信自动通知客户,并发送实验报告查看网址。实验结题后,实验报告、检测结果可在线查看或打印,并永久保留。
文献示例
1、系统进化树结果
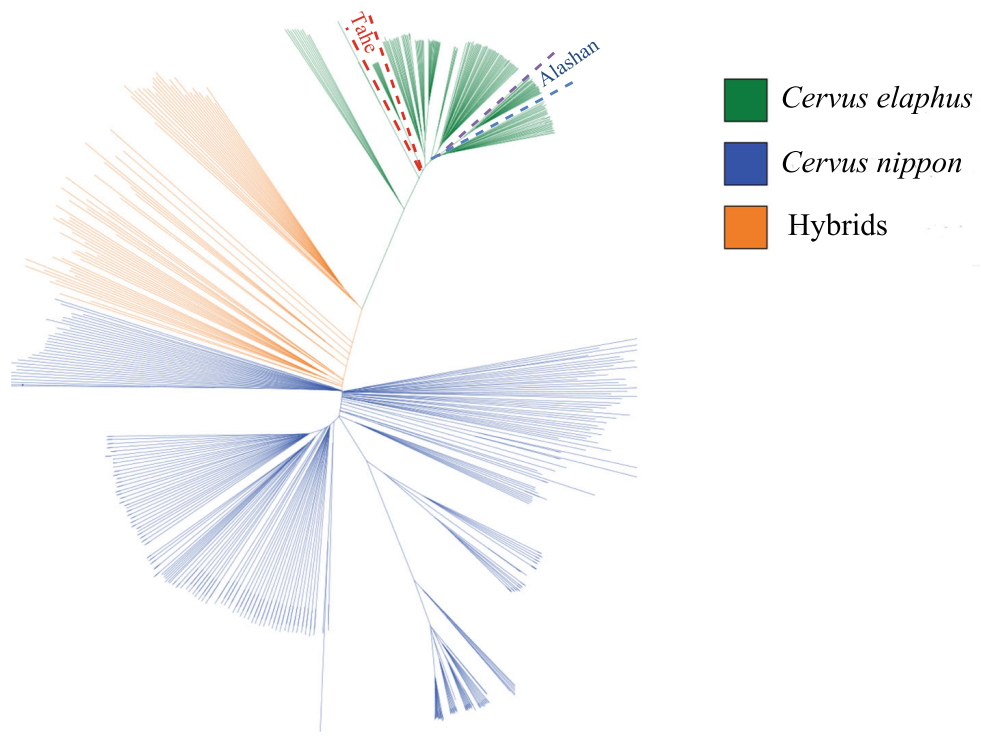
图1 系统进化树分析结果展示(Fan et al, 2021)
2、群体结构分析
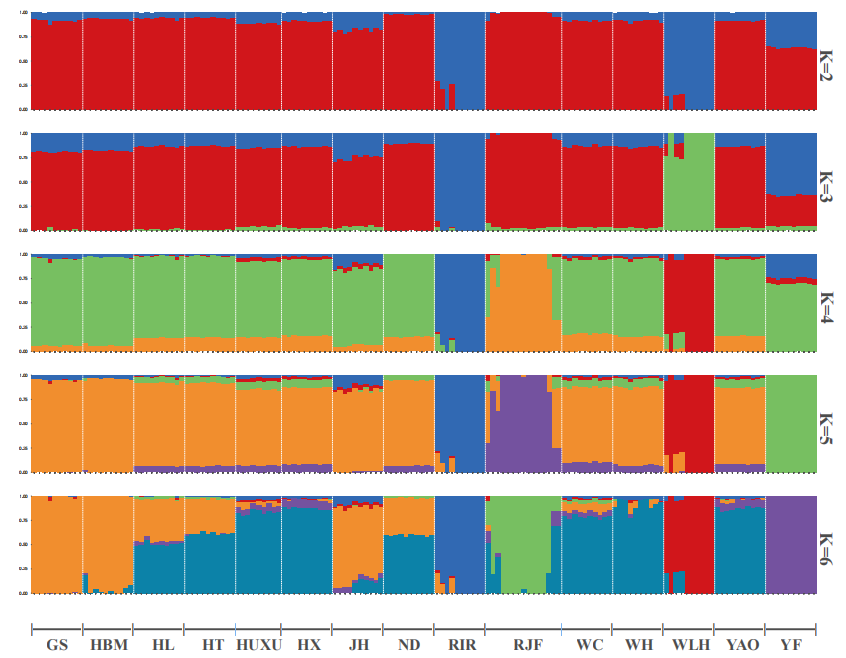
图2 群体结构分析结果展示(Liu et al, 2023)
3、连锁不平衡分析
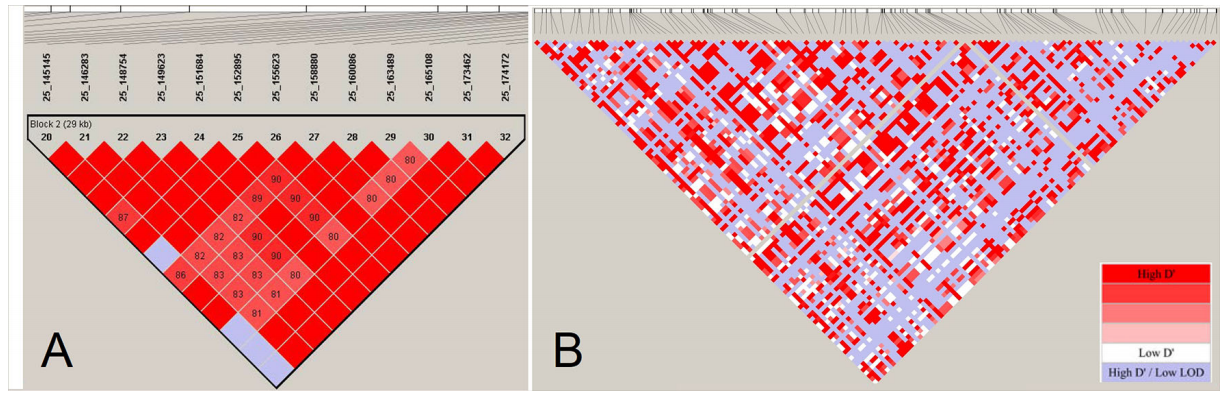
图3 连锁不平衡分析结果展示(Qanbari S, 2020)
4、主成分分析结果
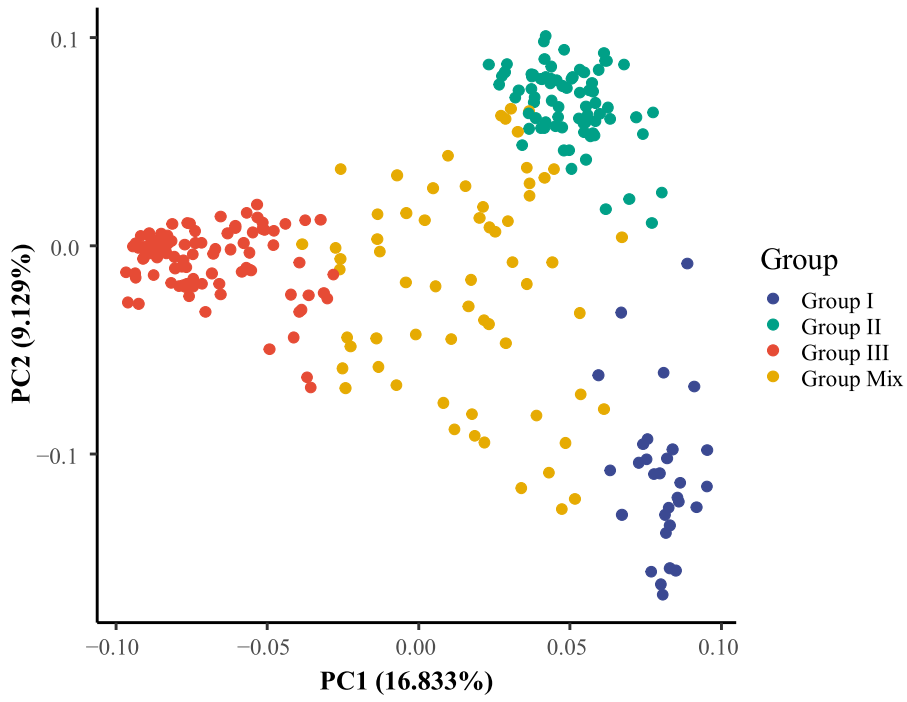
图4 主成分分析结果展示(Chen et al, 2023)
5、单倍型分析结果
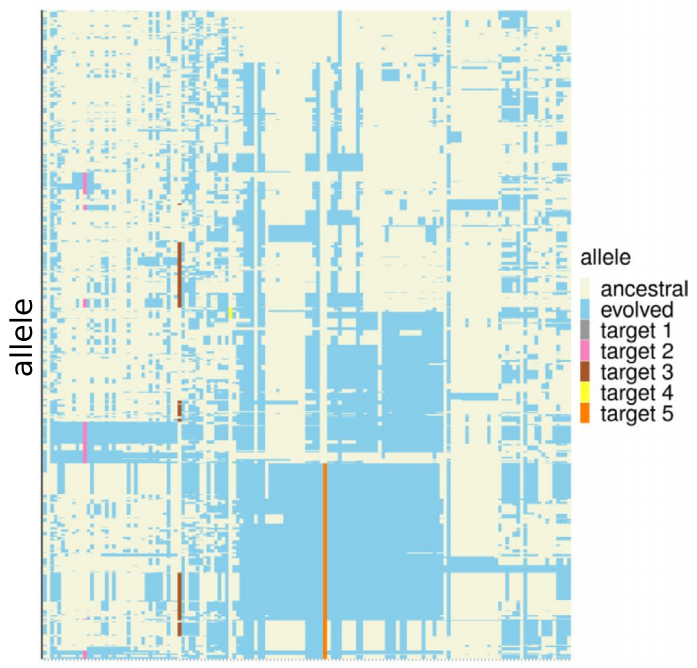
图5 单倍型分析结果展示(Otte et al, 2021)
6、全基因组关联分析结果
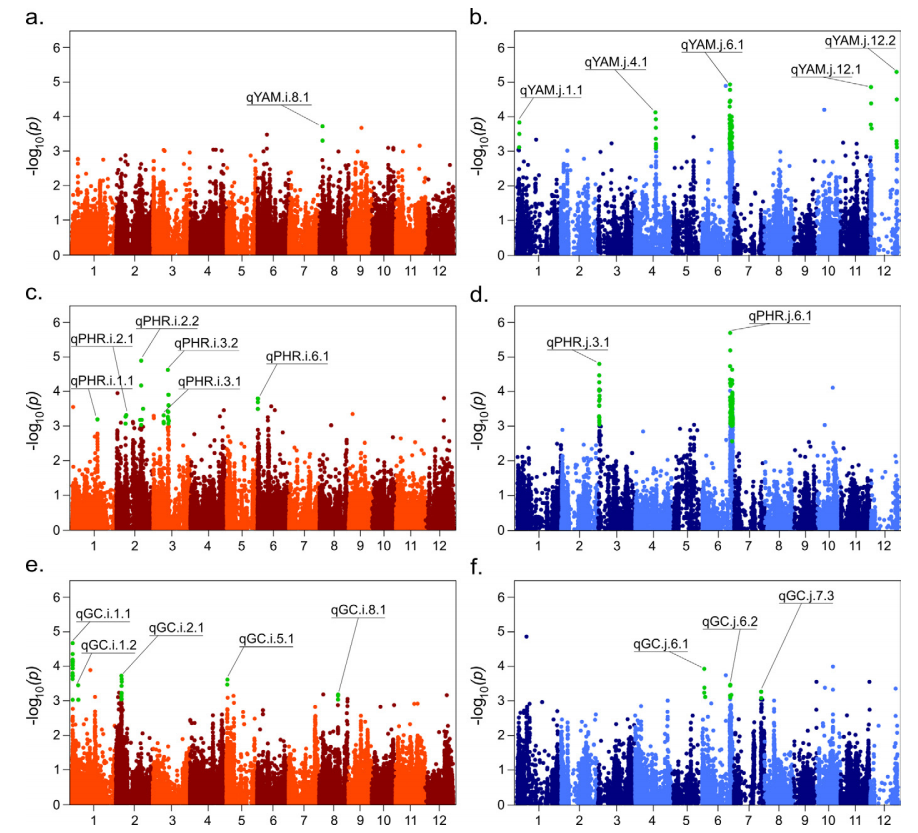
图6 全基因组关联分析结果展示(Quero et al, 2018)
7、eQTL分析
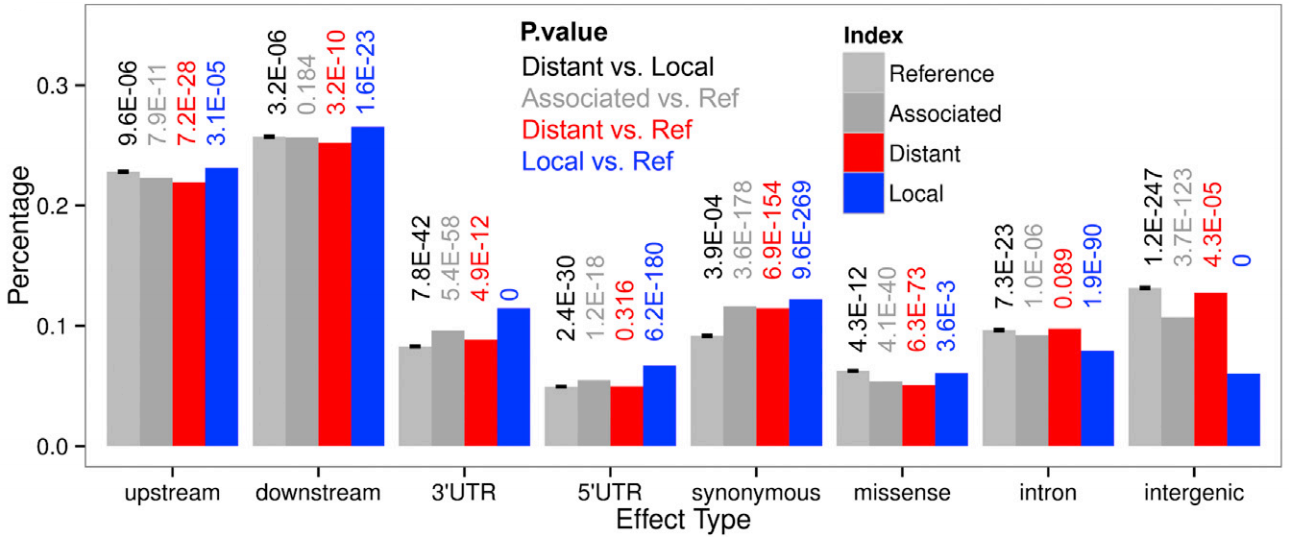
图7 eQTL分析结果展示(Liu et al, 2017)
8、pQTL分析
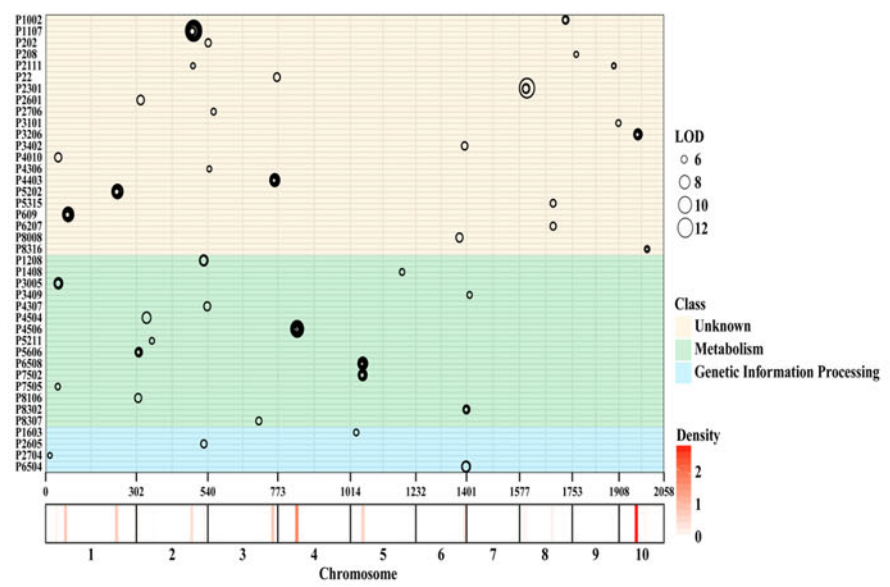
图8 pQTL分析结果展示(Zhou et al, 2021)
9、mQTL分析
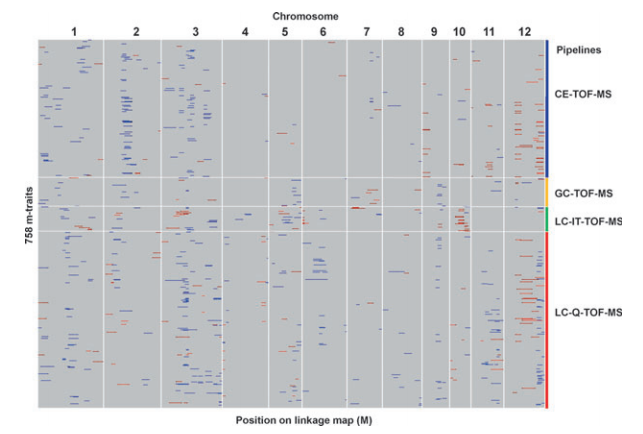
图9 mQTL分析结果展示(Matsuda et al, 2012)
10、HaWAS分析
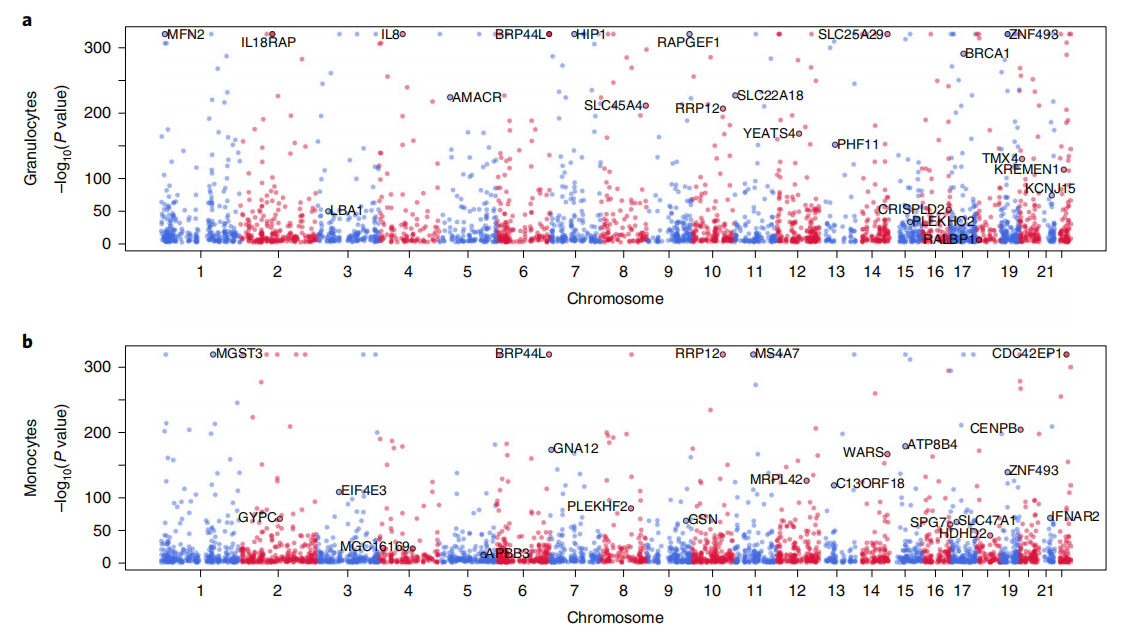
图10 HaWAS分析(Del Rosario et al, 2022
参考文献:
1、Fan H, Wang T, Li Y, et al. Development and validation of a 1 K sika deer (Cervus nippon) SNP Chip[J]. BMC Genomic Data, 2021, 22: 1-13.
2、Liu C, Wang D, He Y, et al. Population structure and genetic diversity analysis of “Yufen 1” H line chickens using whole-genome resequencing[J]. Life, 2023, 13(3): 793.
3、Qanbari S. On the extent of linkage disequilibrium in the genome of farm animals[J]. Frontiers in genetics, 2020, 10: 487295.
4、Chen C, Zhang K, Liu F, et al. Resequencing of global Lotus corniculatus accessions reveals population distribution and genetic loci, associated with cyanogenic glycosides accumulation and growth traits[J]. BMC biology, 2023, 21(1): 176.
5、Otte K A, Schlötterer C. Detecting selected haplotype blocks in evolve and resequence experiments[J]. Molecular ecology resources, 2021, 21(1): 93-109.
6、Quero G, Gutiérrez L, Monteverde E, et al. Genome‐wide association study using historical breeding populations discovers genomic regions involved in high‐quality rice[J]. The plant genome, 2018, 11(3): 170076.
7、Liu, Haijun, et al. "Distant eQTLs and non-coding sequences play critical roles in regulating gene expression and quantitative trait variation in maize."Molecular Plant 10.3 (2017): 414-426.
8、Liu H, Luo X, Niu L, et al. Distant eQTLs and non-coding sequences play critical roles in regulating gene expression and quantitative trait variation in maize[J]. Molecular Plant, 2017, 10(3): 414-426.
9、Matsuda F, Okazaki Y, Oikawa A, et al. Dissection of genotype–phenotype associations in rice grains using metabolome quantitative trait loci analysis[J]. The Plant Journal, 2012, 70(4): 624-636.
10、Del Rosario R C H, Poschmann J, Lim C, et al. Histone acetylome-wide associations in immune cells from individuals with active Mycobacterium tuberculosis infection[J]. Nature Microbiology, 2022, 7(2): 312-326.